- jskm( )を使ってKaplan-Meier生存曲線を描く
- 信頼区間をつける
- 色パレットを変更する
- 線の種類を変更する
- 打ち切りマークを変更する
- Risk tableをつける
- 軸について色々
- 検定結果を表示する
- 凡例を変更する
- 日本語の文字化けを直す
- おわりに
- 参考資料
ggplot系コードで生存曲線を描く方法については以前まとめました。
今回はjskmパッケージを使って生存曲線を描く方法のまとめ。
まずは必要なパッケージを準備する。
library(survival) library(tidyverse) library(jskm)
ここではsurvivalパッケージ内のデータセットpbcを使う。
元々のアウトカム変数statusでは
- 0 = censored
- 1 = transplant
- 2 = dead
となっているが、ここではtrandplantとdeadを合わせた複合アウトカム変数eventを作成して使用する(イベント発生の有無を示す変数は文字列ではダメ)。
> data(pbc) > d <- pbc %>% mutate(event = if_else(status==0,0,1)) > str(d) 'data.frame': 418 obs. of 21 variables: $ id : int 1 2 3 4 5 6 7 8 9 10 ... $ time : int 400 4500 1012 1925 1504 2503 1832 2466 2400 51 ... $ status : int 2 0 2 2 1 2 0 2 2 2 ... $ trt : int 1 1 1 1 2 2 2 2 1 2 ... $ age : num 58.8 56.4 70.1 54.7 38.1 ... $ sex : Factor w/ 2 levels "m","f": 2 2 1 2 2 2 2 2 2 2 ... ~~~ $ event : num 1 0 1 1 1 1 0 1 1 1 ...
jskm( )を使ってKaplan-Meier生存曲線を描く
まずはsurvivalパッケージのsurvfit( )を使って生存時間データに当てはめるモデルを指定する。
fit <- survfit(Surv(time, event)~trt, data = d)
これをjskm( )に渡すだけでよい。
jskm(fit)
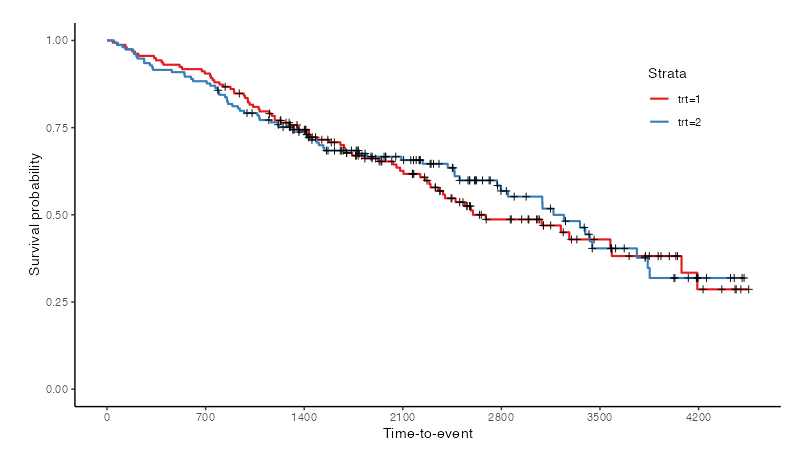
デフォルトでは上記のような生存曲線(survival curve, )が描かれるが、
cumhaz = TRUEを指定することで累積発生関数(cumulative incidence function, )を描くことができる。
ちなみにS(x,t), C(x,t), H(x,t)およびハザード関数には以下のような関係がある。
※ 生存時間にまつわる関数をまとめてみました(2022-07-17 追加)
信頼区間をつける
ci=TRUEを指定すればよい。
jskm(fit, ci = TRUE)

色パレットを変更する
linecolsで色パレットを指定できる。デフォルトはlinecols = "Set1"になっている。白黒で表示したいときは"black"を指定する。
jskm(fit, ci=TRUE, linecols = "black")

白黒にすると自動で線種が点線になるみたい。
線の種類を変更する
dashed = TRUEを指定すると層毎に線種が変更される。
jskm(fit, dashed = TRUE)
出力は割愛。
打ち切りマークを変更する
デフォルトでは打ち切りマークがつく。
付けたくないときはmarks = FALSEと指定する。
jskm(fit, marks = FALSE)
マーカーの種類を指定したいときは、
jskm(fit, shape = "C")
とする(出力割愛)。
Risk tableをつける
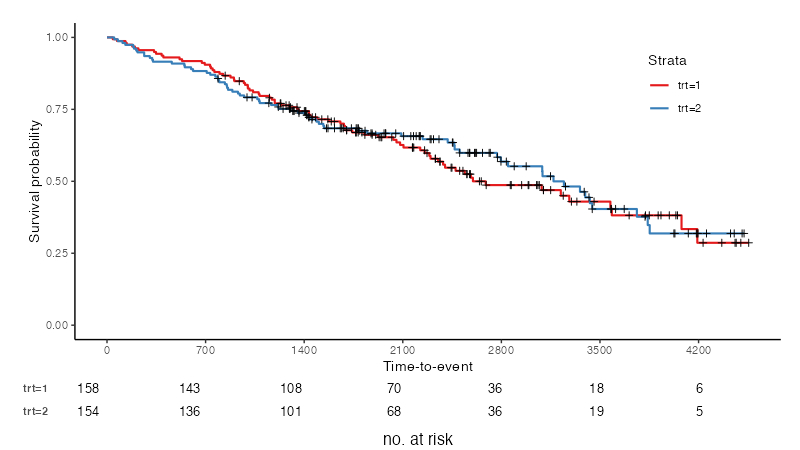
引数table=TRUEと指定すると、at risk人数(number at risk)の表を生存曲線の下につけることができる。
At risk人数表については次の引数も指定できる。
label.nrisk:表のタイトル。デフォルトは"Number at risk"size.label.nrisk:デフォルトは10
jskm(fit, table = TRUE, label.nrisk = "no. at risk", size.label.nrisk = 12)
軸について色々
軸の目盛り間隔を変更する
時間軸の目盛りの間隔は、引数timebyで変更できる(デフォルトは0と最大近くを含めて7つ)。
例えば、時間を500刻みで区切りたいときは、
jskm(fit, timeby = 500)

生存割合の単位をパーセントにしたい場合は、
jskm(fit,surv.scale = "percent")
とする(出力割愛)。
軸の範囲・ラベルを指定する
軸の範囲はxlims, ylimsで指定する。
jskm(fit, xlims = c(0,2000))
軸のラベルはxlabs, ylabsで指定できる。ちなみに図のメインタイトルはmainで指定できる(出力割愛)。
検定結果を表示する
図内にP値を表示したい場合はpval = TRUEを指定する。他に指定できる引数は、
pval.size:フォントサイズ(デフォルトは5)pval.coord:表示位置pval.testname:検定名を表示するかどうか
がある。
jskm(fit, data=d, pval = TRUE, pval.size = 6, #デフォルト=5 pval.coord = c(700,0.5), pval.testname = TRUE)

凡例を変更する
凡例を表示するときはlegend = TRUEを指定する。凡例については他に以下の引数を指定できる。
legendposition:表示場所。両軸ともに0から1の範囲で指定する。ystrataname:凡例のタイトルystratalabs:凡例の層ラベル
jskm(fit, legend = TRUE, legendposition = c(0.2,0.5), ystrataname = "Treatment", ystratalabs = c("Treatment A", "Treatment B"))

日本語が白い四角に文字化けしてしまう場合は次を参照。
日本語の文字化けを直す
Macでは日本語が豆腐化することがある。
そのようなときは、RStudioの設定を以下のように変更する。
Preference→Graphicと進み、BackendをAGGに変更する。- raggパッケージをインストールするか聞かれるので、インストールする。
おわりに
- survminerより指定できることは少ないが、シンプルで使いやすいかも。
cut.landmarkを使うとランドマーク解析の生存曲線が描ける。- こたつに足を突っ込んで、猫を蹴ってしまい、猫より人間の方がびっくりすることがよくあります。
参考資料
- 開発者のvignetteページ: