- ggsurvplot( )を使ってKaplan-Meier生存曲線を描く
- 信頼区間をつける
- 色パレットを変更する
- 線の種類を変更する
- 打ち切りマークを変更する
- 中央値を示す補助線をつける
- Risk tableをつける
- 軸について色々
- 凡例を変更する
- 日本語の文字化けを直す
- おわりに
- 参考資料
※ 他の関数を使って生存曲線を描く記事も書きました(2022-07-17 追加)
基本の作図関数plot( )ではなくsurviminerパッケージを使って生存曲線を描く方法のまとめ。
まずは必要なパッケージを準備する。
library(survival) library(survminer) library(tidyverse)
ここではsurvivalパッケージ内のpbcデータセットを使う。
もともとイベント発生状況を示す変数statusでは
- 0 = censored
- 1 = transplant
- 2 = dead
となっているが、ここではtrandplantとdeadを合わせた複合アウトカム変数eventを作成して使用する(イベント発生の有無を示す変数は文字列ではダメ)。
> data(pbc) > d <- pbc %>% mutate(event = if_else(status==0,0,1)) > str(d) 'data.frame': 418 obs. of 21 variables: $ id : int 1 2 3 4 5 6 7 8 9 10 ... $ time : int 400 4500 1012 1925 1504 2503 1832 2466 2400 51 ... $ status : int 2 0 2 2 1 2 0 2 2 2 ... $ trt : int 1 1 1 1 2 2 2 2 1 2 ... $ age : num 58.8 56.4 70.1 54.7 38.1 ... $ sex : Factor w/ 2 levels "m","f": 2 2 1 2 2 2 2 2 2 2 ... ~~~ $ event : num 1 0 1 1 1 1 0 1 1 1 ...
ggsurvplot( )を使ってKaplan-Meier生存曲線を描く
まずはsurvivalパッケージのsurvfit( )を使って生存時間データに当てはめるモデルを指定する。
fit <- survfit(Surv(time,event)~trt, data = d)
これをggsurvplot( )に渡すだけでよい。
ggsurvplot(fit)
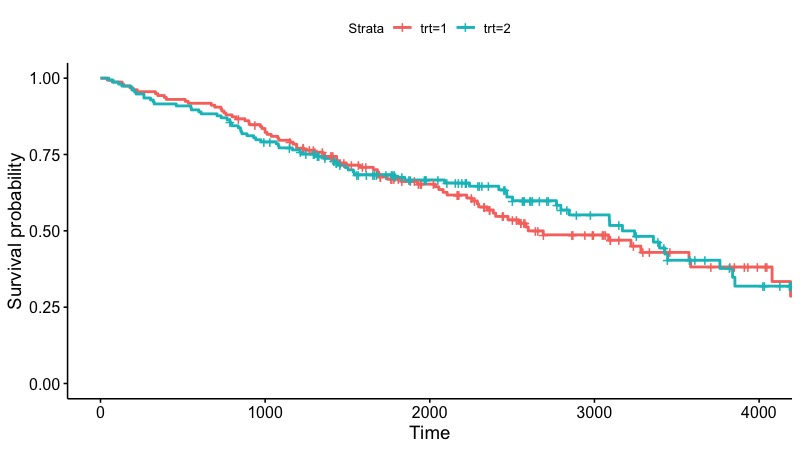
デフォルトでは上記のような生存曲線(survival curve, )が描かれるが、
funを指定することで以下の関数を描くことができる。
fun="event":累積発生関数(cumulative incidence function,)
fun="cumhaz":累積ハザード関数(cumulative hazard function,)
ちなみにS(x,t), C(x,t), H(x,t)およびハザード関数には以下のような関係がある。
※ よく出てくるのでまとめ直しました(2022/7/17 追加)
全体の生存曲線を重ねたい場合はadd.all=TRUEを指定する。
ggsurvplot(fit, add.all=TRUE)
信頼区間をつける
conf.int=TRUEを指定すればよい。
信頼区間については他にも以下の引数を指定可能:
conf.int.style:"step"とすると境界線のみ、"ribbon"とすると帯を使って描く。デフォルトは後者。conf.int.alpha:透過性を0(完全透過)〜1(完全不透過)で指定。
ggsurvplot(fit,conf.int=TRUE)

色パレットを変更する
Rでは様々なパレット(色の組み合わせ)が用意されている。
以下のサイトが詳しい。
特にggsciパッケージでは科学雑誌別の色パレットを提供しているので便利。
例えばLancetを指定すると、
library(ggsci) ggsurvplot(fit, palette="lancet")
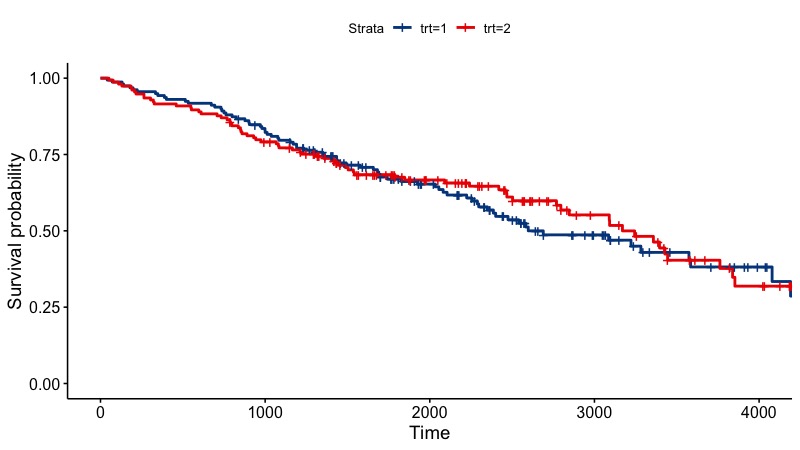
ちなみに、それぞれの群について色を指定する場合も引数paletteで指定する(結果割愛)。
ggsurvplot(fit, palette=c("magenta", "green"))
線の種類を変更する
引数linetypeで指定する。
solidまたは1:実線dashedまたは2:破線dottedまたは3:点線dotdashまたは4:一点鎖線longdashまたは5:長い破線twodashまたは6:長短交互の破線blankまたは0:何も描かない
各線がどのように見えるかは以下のサイトを参照。
ggsurvplot(fit, linetype=c("solid","dotted"))
打ち切りマークを変更する
デフォルトでは打ち切りマークがつく。
付けたくないときはcensor=FALSEと指定する。
他には以下の引数が指定できる。
censor.shape:打ち切りマークの種類を指定。デフォルトは"+"censor.size:マーカーの大きさを指定。デフォルトは4.5
中央値を示す補助線をつける
引数surv.median.lineで中央生存期間に関する補助線を引く。
"h":生存確率=0.5の水平な線のみ"v":各群の中央生存期間を示す垂直線のみ"hv":上記の両方
ggsurvplot(fit, surv.median.line=c("hv"))

Risk tableをつける
引数risk.table=TRUEと指定すると、at risk人数(number at risk)の表を生存曲線の下につけることができる。
引数risk.tableには他にも次のような値を指定できる。
"percentage":at risk人数の代わりにat risk症例の割合を示す"abs_pct":at risk人数の後に、パーセンテージを付記"nrisk_cumcensor":at risk人数の後に、累積打ち切り人数を付記"nrisk_cumevents":at risk人数の後に、累積イベント発生人数を付記
ggsurvplot(fit, risk.table="abs_pct")
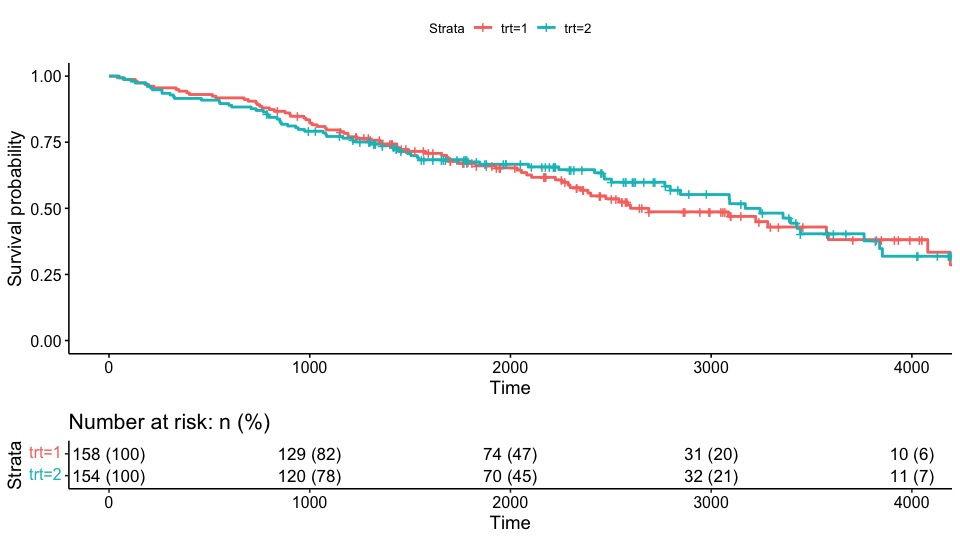
Risk tableを付ける場合、以下の引数で書式変更可能。
tables.height:表の高さ。0-1の範囲で指定。risk.table.title:表のタイトルrisk.table.pos:表の場所。デフォルトは"out"で、"in"とすると生存曲線のグラフ領域内に表が追加される。
軸について色々
軸の目盛り間隔を変更する
軸の目盛りの間隔は、引数break.x.byおよびbreak.y.byで変更できる。
x軸についてはbreak.time.byとしてもOK。
例えば、x軸は250刻み、y軸は0.1刻みで区切りたいときは、
ggsurvplot(fit, break.x.by=500, break.y.by=0.1)

軸の単位を変更する
引数xscaleを指定することで、時間の単位を変更できる。
例えば、元々が日単位のものを、年単位に変更したいときはscale="d_y"と指定する。
月単位にしたいときはmを使う。
生存割合の表示はデフォルトでは0-1で表記されているが、パーセント表示にしたいときはsurv.scale="percent"と指定すればよい。
軸の範囲を指定する
xlim, ylimで指定する。
凡例を変更する
凡例については以下の引数を指定できる。
legend:表示場所。"right", "left", "top", "bottom", "none"の他、全体に対する割合を使ってc(0.8, 0.8)のように指定もできる(例えば右上にしたいとき、"topright"は使えないので後者を使う必要がある)。legend.title:凡例のタイトルlegend.labs:凡例のラベル
凡例と合わせて、グラフタイトルや軸ラベルも変更してみる。
ggsurvplot(fit, title= "メインタイトル", xlab="時間", ylab="生存割合", legend=c(0.8,0.8), #"top"/"bottom"/"left"/"right"/"none"またはc(x,y)でx,y=0-1を指定 legend.title="凡例", legend.labs=c("治療1","治療2"))
日本語が白い四角に文字化けしてしまう場合は次を参照。
日本語の文字化けを直す
Macでは日本語が豆腐化することがある。
そのようなときは、RStudioの設定を以下のように変更する。
Preference→Graphicと進み、BackendをAGGに変更する。- raggパッケージをインストールするか聞かれるので、インストールする。
上手くいくと下のように表示される。

おわりに
- ggplotはキレイな色パレットが使えるので重宝しますね。
参考資料
- 開発者の説明: rpkgs.datanovia.com