ランドマーク(LM)解析とは
Immortal time biasなど時間依存性変数によって生じる問題に対処するための手法の1つ。臨床的観点から妥当なLM時点(landmark time-point)を設定して、以下の要領でat risk集団と要因・介入の状態を判定する。
- LM時点でイベントを発生していない人がat risk集団として解析対象となる
- LM時点前に要因・介入が発生した場合に「要因・介入あり」と判定する
- LM時点後に要因・介入が発生しても「要因・介入なし」と判定する
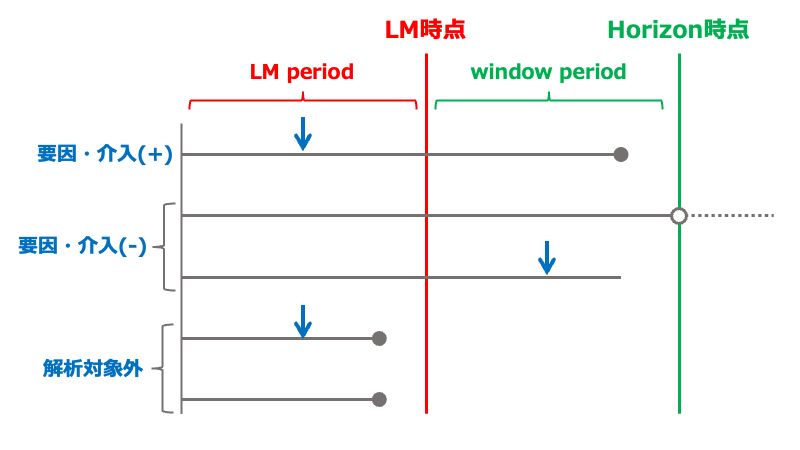
LM時点の他にも、強制的に打ち切りとする時点(horizon)が設定されることがある。これによって、LM時点まで生存している条件下で、LMからhorizonまでの期間(window)で予測される生存確率を推定できる。
シンプルなランドマーク解析については以前まとめた。
スーパーランドマーク解析とは
どの時点をLMに選ぶかによって解析結果は変わるので、臨床的に妥当と考えられる時点を選んだり、感度解析として複数のLM時点で解析をしたりする方がよい。複数のLM時点の結果を層別解析として統合する「スーパーランドマーク解析*1」と呼ばれる方法がある。
必要なパッケージとデータの読み込み
dynpredパッケージの他、データハンドリングのためにtidyverseパッケージとmagrittrパッケージを読み込んでおく。
magrittrパッケージは、引用元を上書きしてくれるパイプ記号%<>%を使うために必要。
library(dynpred) library(tidyverse) library(magrittr)
dynpredパッケージに含まれるwbc1, wbc2データを使う。 このデータは、慢性骨髄性白血病(CML)患者のアウトカムと時間依存性共変量としてWBC値を含んでいる*2。
それぞれのデータに含まれる変数は以下のとおり。
wbc1データ
patnr:患者IDtyears:登録から死亡または最終観察までの期間(年)d:1=生存, 0=打ち切りsokal:脾臓サイズ、芽球割合、血小板数、年齢から算出される臨床スコアage:登録時年齢
wbc2データ
patnr:患者IDtyears:登録からWBC測定時点までの期間(年)lwbc:WBC数の対数値
tyearsという変数名が重複してしまってややこしいので、生存期間はsurvtime、WBC測定までの期間はwbctimeに変更する。2つのデータを結合して、dataという名前で保持する。
data(wbc1) data(wbc2) wbc1 %<>% rename(survtime = tyears) wbc2 %<>% rename(wbctime = tyears) data <- wbc1 %>% left_join(wbc2, by = "patnr")
こんな感じの縦長データができた。
> head(data, n = 20) patnr survtime d sokal age wbctime lwbc 1 1 1.308333 1 0.66 57.3 0.07671233 -0.0435092748 2 1 1.308333 1 0.66 57.3 0.11506849 -0.1518487496 3 1 1.308333 1 0.66 57.3 0.15342466 -0.1039251973 4 1 1.308333 1 0.66 57.3 0.19178082 -0.0666771399 5 1 1.308333 1 0.66 57.3 0.22191781 -0.0215149811 6 1 1.308333 1 0.66 57.3 0.25205479 -0.0109219076 7 1 1.308333 1 0.66 57.3 0.33698630 0.0290413923 8 1 1.308333 1 0.66 57.3 0.41369863 0.0828372247 9 1 1.308333 1 0.66 57.3 0.50958904 -0.0974910873 10 1 1.308333 1 0.66 57.3 0.59452055 -0.0109219076 11 1 1.308333 1 0.66 57.3 0.65205479 -0.0005810743 12 1 1.308333 1 0.66 57.3 0.76712329 -0.0215149811 13 1 1.308333 1 0.66 57.3 0.85479452 0.3644662262 14 1 1.308333 1 0.66 57.3 0.90136986 0.5171580313 15 1 1.308333 1 0.66 57.3 1.00547945 0.9848718175 16 2 4.710000 1 0.99 19.9 0.01917808 -0.5150266520 17 2 4.710000 1 0.99 19.9 0.03835616 -0.3617982759 18 2 4.710000 1 0.99 19.9 0.07945205 -0.4114860601 19 2 4.710000 1 0.99 19.9 0.13424658 -0.3172161433 20 2 4.710000 1 0.99 19.9 0.17260274 -0.3502164034
使用するスーパーランドマーク解析の概要
下の図のように、1人の患者に対して複数回にわたってWBCが計測されている。 ここで、次の要領でランドマーク解析を行い、結果を統合する。
- LM時点を0.5年から3.5年まで0.1年刻みでスライドさせていく。
- window期間は4年とする。
- LM時点の直近0.25年に計測されたWBC値のみ採用する(時間的に離れているものは除外)。
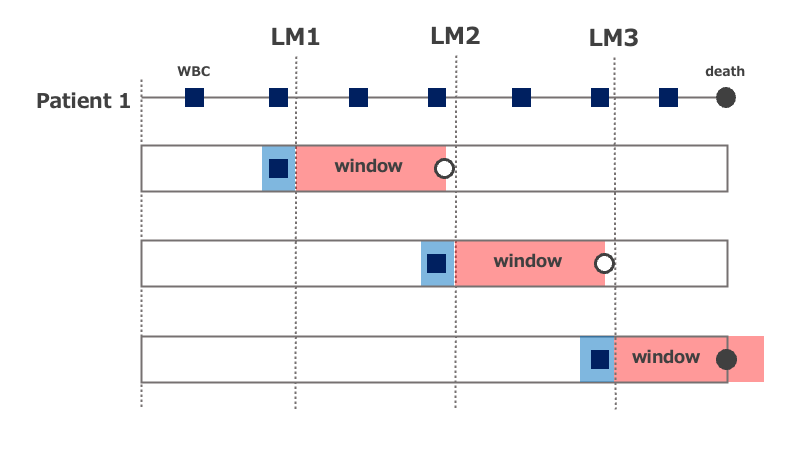
上の例では、LM1, LM2のときはwindow期間内にアウトカムが観察されていないので「打ち切り」として扱われる。LM3の時はwindow期間内にアウトカムが観察されている。
参考書籍では、次の3つのモデルを使って解析していたが、今回はひとまずモデル1だけやってみる。
- LMを層とした層別Cox回帰モデル(window期間内でWBCの係数は一定)
- WBC数の効果について時間依存性を考慮した層別Cox回帰モデル(モデル1 + time-varying coefficient)
- ベースラインハザードをLMの関数としたCox回帰モデル
cutLM( )を使ってLM解析用データセットを準備する。
cutLM( )を使ってLM解析用のデータセットを作る。cutLM( )の詳細は以前の記事を参照のこと。
LM時点をLMsとして決めて、これを変数にもったデータフレーム*3を作成し、それぞれのLM時点に対してcutLM( )で作成したデータフレームを割り当てる(purrrパッケージのmap( )を使用)。最後はbind_rows( )を使ってそれを全て縦につなげる。
LMs <- seq(0.5, 3.5, by=0.1) LM_tibble <- tibble(LM = LMs) fun <- function(LM){ d <- cutLM(data = data, #dataはwbc1, wbc2から作ったもの outcome = list(time="survtime",status="d"), LM = LM, horizon = LM+4, covs = list(fixed=c("sokal","age"),varying="lwbc"), format = "long", id = "patnr", rtime = "wbctime") %>% filter(LM - wbctime <= 0.25) #LM前0.25年超は除外 return(d) } LM_tibble %<>% mutate(LM_data = map(LM, fun)) superLM_data <- LM_tibble$LM_data %>% bind_rows()
これを、LMを層としたCoxモデルに投入。同一患者で複数の観察が含まれるので、cluster( )で指定してロバスト分散を用いる。
superLMcox1 <- coxph(Surv(survtime,d) ~ age + sokal + lwbc + strata(LM) + cluster(patnr), data = superLM_data , method="breslow")
結果はこちら。
> superLMcox1 Call: coxph(formula = Surv(survtime, d) ~ age + sokal + lwbc + strata(LM), data = superLM_data, method = "breslow", cluster = patnr) coef exp(coef) se(coef) robust se z p age 0.011116 1.011178 0.002233 0.009859 1.128 0.25952 sokal 0.581892 1.789421 0.051347 0.193969 3.000 0.00270 lwbc 0.819992 2.270482 0.086056 0.255964 3.204 0.00136 Likelihood ratio test=267.8 on 3 df, p=< 2.2e-16 n= 3641, number of events= 1409
参考書籍の結果と一致していたのでOKとする。
おわりに
- 解析そのものよりは、データセットの準備と結果の解釈の方が難しいと思いました。
- 冷蔵庫で冷えたウェットタイプの餌を、お湯でほぐしてあげると食べる勢いがすごいです。
参考資料
- Landmark解析のほか、multi-state modelを使った生存時間の動的予測について書かれています(ハードカバー版はお値段高めだったのでKindle版にしました)。スーパーランドマークについてはChapter 8です。
*1:「スーパーランドマークモデルを使った解析」が適切か。
*2:Kantarjian HM, Talpaz M. Questions raised by the Benelux CML Study Group: results from the randomized study with hydroxyurea alone versus hydroxyurea combined with low-dose interferon-alpha2b for chronic myeloid leukemia. Blood. 1998 Oct 15;92(8):2984-7. PMID: 9763593.
*3:正確にはtibble

